NGS样品准备
导语
RNA的结构特征决定了其半衰期短,容易降解的特性,对RNA来源样品的正确采集取样和运送操作,可以有效降低RNA降解的风险。转录组测序中,常见的样品类型包括:动物组织样品、植物组织样品、原核生物样品以及抽提好的Total RNA等;如下,本文会分别根据不同类型的情况详细描述各类型样品的特征以及采集RNA样品的注意事项。
组织在离体后会迅速释放Rnase,会大范围的降解RNA,再加上RNA的半衰期较短,因此RNA在离体组织中会迅速降解,但借助低温条件(-40℃以下)可以有效保存RNA以及抑制Rnase的活性。
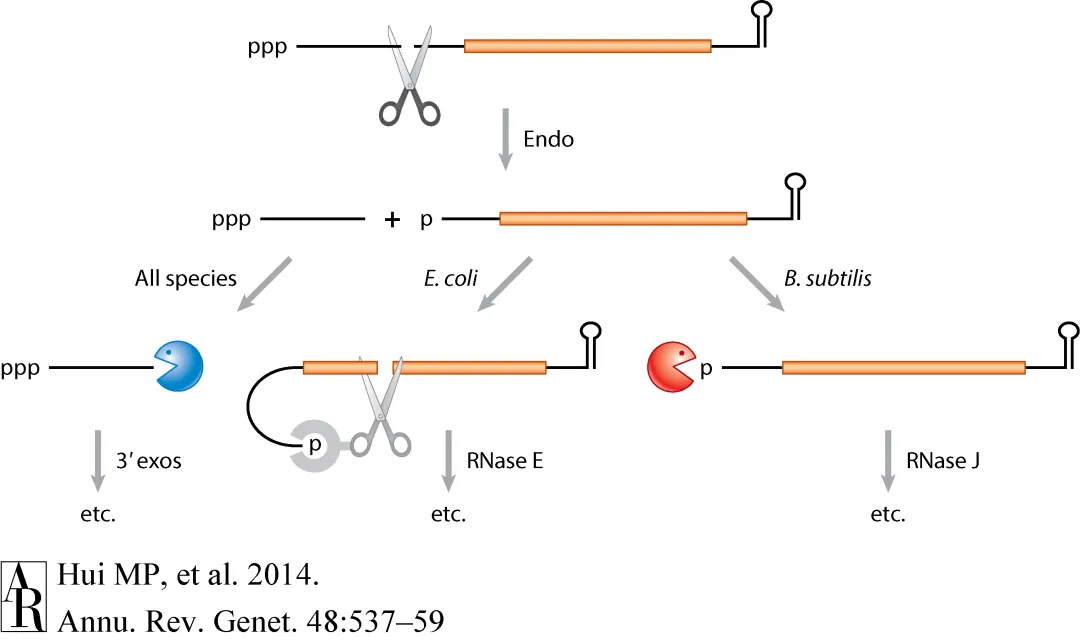
RNA降解
整体样品量建议
(1)RNA样品要求
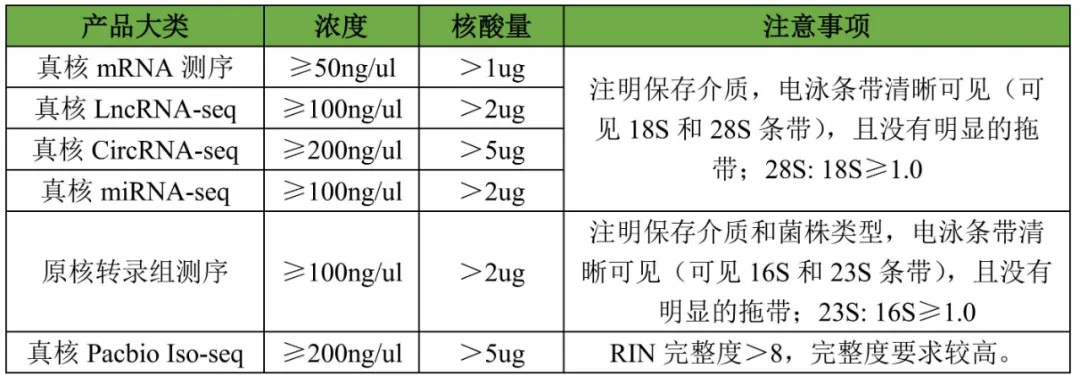
(2)组织样品要求
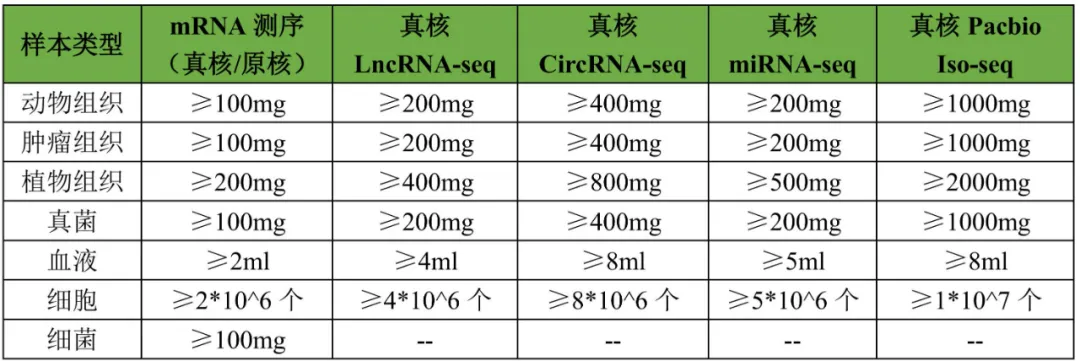
注:如果是全转录组项目(同时做lncRNA-seq、miRNA-seq和circRNA-seq),上述单项产品样品量相加即可。
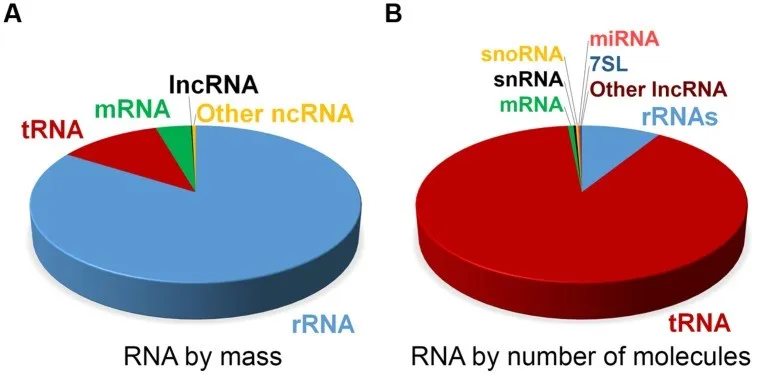
Total中RNA类型和质量占比及数量占比
注:由于Total RNA中含有大量的结构RNA(rRNA等)和其他调控性RNA,用于研究的mRNA和各种ncRNA(lncRNA、miRNA、circRNA)的占比较低,因此转录组测序项目的样品需求量相对较多。
取样准备
(1)器材准备
试剂耗材准备:双蒸水、预冷的PBS、无水乙醇、液氮、干冰(根据运送距离定,一般为10-15kg),无Rnase的离心管、锡箔纸、一次性乳胶手套、一次性口罩等。
工作台准备:超净工作台、解剖剪、镊子、手术刀;上述器具和平台均需要进行灭菌和无Rnase处理。
运送准备:保存采集好样品的离心管的时候,需要准备质体柔软的包材(泡沫板)或形成独立空间的大体积离心管(50ml、100ml离心管),以保证运送过程中离心管管的完整。
(2)工作准备
1.根据指定好的研究方案,选取合适的取样材料和取样时间,如果是要分批次采集的话,需要规划好材料的前期准备,同时分批采集的样品可以先放置在-80℃,后续一次性通过干冰保存送样(防止不同批次测序的批次效应)。
2. 无Rnase的离心管标记好编号或送样名称(只要能跟送样单上的名称对应即可),标记好之后进行液氮预冷(防止后续速冻样品加入,由于温度差导致炸管)。
3. 操作前再对已经灭菌操作的器具使用酒精擦拭,防止外源RNA的污染。

样品采集细则
样品采集细则部分,针对动物组织、植物组织、贴壁细胞、悬浮细胞、血液、原核细胞等常见样品类型进行汇总和采集细则注意事项的阐述,希望可以给各位老师带来一点帮助,以便于更好地根据采集到的样品分离纯化RNA,进行后续的RNA文库构建和测序。
(1)动物组织取样流程
1. 动物活体取材后,立即使用Rnase-free水进行清洗后在冰上迅速进行后续操作:剔除结缔组织和脂肪组织等非研究部位,之后目标组织被切割成黄豆大小(50mg-100mg)的组织块。
2. 切割好的组织块,液氮速冻后放入预冷的EP管中,干冰寄送;放置到-80℃冰箱中保存。
(2)植物组织取样流程
1.选定植物组织部位(根、茎、叶等),采集前先采用PBS/Rnase-free水擦拭或冲洗干净,再用吸水纸吸干样品表面。
2. 切割要采集的植物组织,将组织切割成绿豆大小的小块(50-100mg),切割过程在冰上迅速处理,之后进行液氮速冻(10min以上,保证充分速冻)并放入预冷好的EP管中。注:一般会多采集几份,一部分用作备份。
3. 上述包含速冻好组织的EP管,备份部分放置到-80℃冰箱中保存。
(3)贴壁细胞取样流程
1. 倒掉培养瓶中的培养液,并加入适量的PBS溶液进行清洗(2-3次),最后一次清洗的时候不倒掉PBS,使用细胞刮勺刮取细胞,将细胞连通PBS溶液一起收集到EP管中,500g离心3-5min,丢弃上清,收集沉淀细胞。
2.在收集到的离心沉淀细胞中,每1*10^6个细胞加1ml Trizol,适用枪头充分吹打或轻轻震荡,使得细胞充分裂解,冰上低温孵育5min(保证Trizol使得Rnase完全变性),转移到干冰中进行寄送。备份部分转移到-80℃低温保存。
(4)悬浮细胞取样流程
1. 将悬浮的细胞连同培养基一起倒入50ml/100ml离心管中,500g离心3-5min,弃去培养液,之后进行2-3次的PBS清洗,离心操作同上弃去上清,最后收集沉淀细胞。
2. 在收集到的离心沉淀细胞中,每1*10^6个细胞加1ml Trizol,适用枪头充分吹打或轻轻震荡,使得细胞充分裂解,冰上低温孵育5min(保证Trizol使得Rnase完全变性),转移到干冰中进行寄送。备份部分转移到-80℃低温保存。
(5)血液样品取样流程
1.全血样本比较特殊:全血样本中含有大量的globinmRNA转录本(globinmRNA转录本主要存在于成熟红细胞中,其序列和功能保守,与rRNA类似),globinmRNA转录本会占据大量的数据,使得有效转录本数据量降低,显著降低对其低丰度转录本检出的敏感性;因此建议样品采集时,先分离白细胞再提取RNA。
2. 使用EDTA抗凝管收集血液,然后加入等体积的1×PBS,充分混匀;然后将上述混匀好的血液,缓慢转入另一加入淋巴细胞分离液的离心管中,并使上述混合液处于淋巴细胞分离液液面之上(即两种液体不要混合,保留清晰的接口),3000g离心30min。
3. 用移液器小心分离出白细胞层,用PBS(1×)清洗白细胞,离心回收白细胞,弃去上清;按照2ml血液分离白细胞或10^6白细胞对应1ml Trizol的比例加入适量Trizol,用吸头吹打或轻轻震荡,使得细胞充分裂解,冰上低温孵育5min(保证Trizol使得Rnase完全变性),转移到干冰中进行寄送。备份部分转移到-80℃低温保存。
注:如果没有分离白细胞条件,针对于全血的操作是:每1ml血液加入3mlTrizol的比例加入适量Trizol,用吸头吹打或轻轻震荡,使得细胞充分裂解,冰上低温孵育5min(保证Trizol使得Rnase完全变性),转移到干冰中进行寄送。备份部分转移到-80℃低温保存。
EDTA抗凝管
微生物培养液取样流程
1. 选取大于OD500或者达到对数中后期的菌液(严格控制非变量条件的一致性),在4℃预冷的离心机中10000-12000×g离心3-5 min沉降菌体于50 ml离心管中。留少量菌液,将菌体重悬(全程需无菌操作)。对于厌氧细菌,除按如上操作外,在培养与菌体收集过程中需严格控制厌氧条件。
2. 重悬后(只留取少量菌液),转移至2ml圆底离心管(便于取出进行低温研磨),液氮速冻,转移至干冰寄送;备份部分转移到-80℃低温保存。
微生物培养基取样流程
除去培养基,收集菌体,之后迅速液氮速冻,干冰寄送;备份部分转移到-80℃低温保存。
注:虽然微生物样品类型与细胞样品类型有其相似性,但微生物样品一般不使用Rnalater保存或者Trizol裂解液裂解后送样;原因是Rnalater中的微生物样品由于Rnalater密度大而不好进行分离,同时由于大多数微生物均有比较复杂的细胞壁结构,因此裂解前可能需要进行低温研磨处理以便更好地释放出RNA;综上,不建议微生物样品使用Rnalater或Trizol进行保存操作。
样品包装和寄送
1. 用1.5ml或2ml离心管保存和寄送,建议将样品管盖用封口膜缠绕4-5圈。
2. 所有组织样品均建议离体后经简单清洗后立即液氮速冻,然后-80℃保存。
3. 寄送过程中,干冰保证稳定的低温环境,将PE管包裹在泡沫板中,防止运输过程中的颠簸使得PE管破碎,样品泄露(低温下,PE管更容易破碎)。

注意事项
(1)取样原则
1.代表性:准确选取课题研究部分(肿瘤核心、感病区间等),剔除与研究无关的组织部位(结缔组织、共生物种组织等)
2. 一致性:保证实验组和对照组样品采集部位和时间的一致性,在研究条件之外的其他指标要尽可能保持一致。
3. 迅速性:采集、制备、贮存、运输过程中应尽可能地做到迅速,最大限度的缩短从样本采集到实验的时间。
4. 简洁性:样品命名要简洁,一般元素为大小写英文字母、下划线_和数字;简洁的命名便于书写在EP管上,同时便于实验员记录和管理样品,以及后续数据分析的时候也就不需要频繁的更改名字以符合LINUX系统上运行的RNA-seq分析流程的命名标准。
(2)组织类型建议&特殊组织
1. 动物组织:建议采集含有高丰度RNA的组织(肝脏、胸腺、胰腺、脾脏、脑、肌肉等组织),如果要采集特殊组织部位,需在送样前跟驻地销售沟通,明确特殊组织可能存在的风险,例如:软骨组织抽提RNA的效率很低且容易降解,脂肪组织由于其富含的脂肪会严重干扰RNA的抽提。
2. 植物组织:建议采集含有高丰度RNA的组织(发育期的根、茎和叶),同时避免一些共生组织的采集(除非研究的就是共生组织,例如根瘤);植物表面的蜡质需要尽可能擦除;同时特殊样品(如多糖多酚多优质)需联系销售备注,便于实验室采用对应的试剂盒抽提。
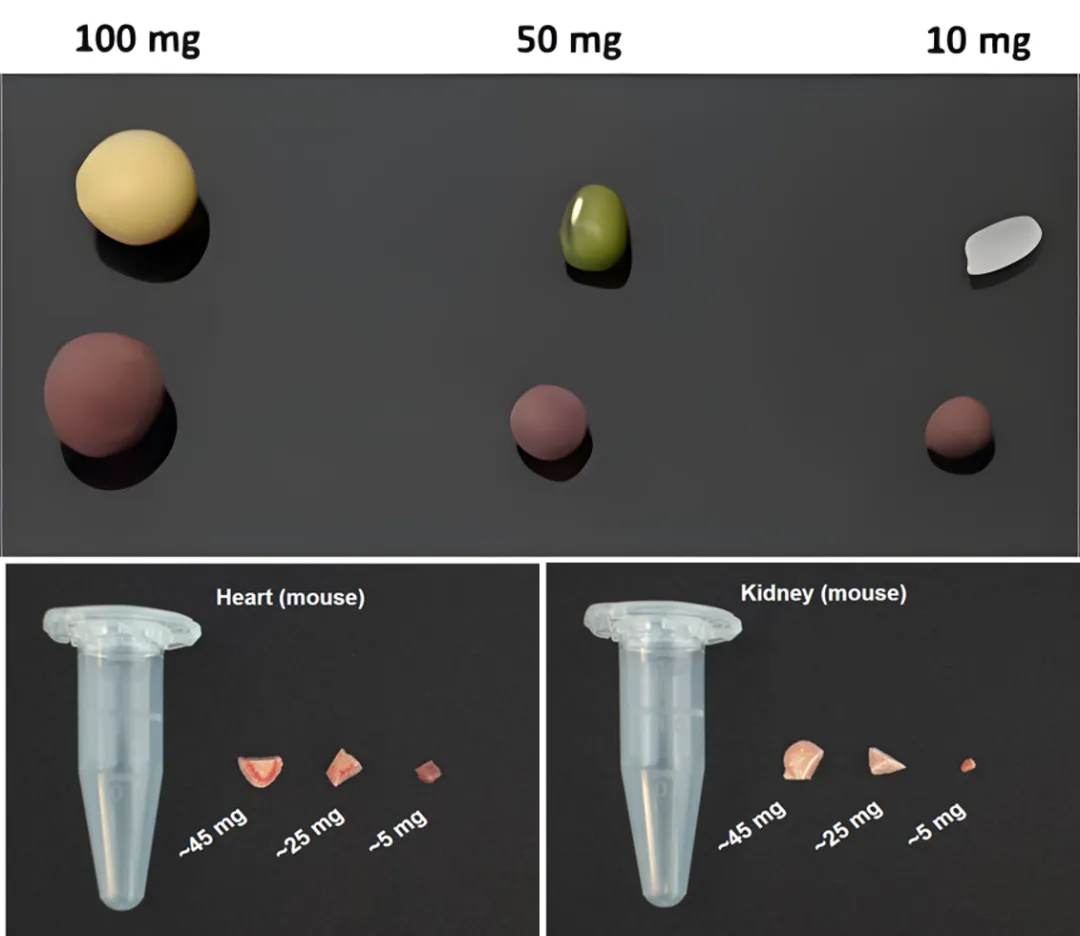
(3)样品重量
如果为常规组织大小的样品重量参考示意图
参考文献
1.Trivedi, Christopher B et al. “DNA/RNA Preservation in Glacial Snow and Ice Samples.” Frontiers in microbiology vol. 13 894893. 23 May. 2022, doi:10.3389/fmicb.2022.894893.
2.Houseley, Jonathan, and David Tollervey. "The many pathways of RNA degradation." Cell 136.4 (2009): 763-776.
3.Hui, Monica P., Patricia L. Foley, and Joel G. Belasco. "Messenger RNA degradation in bacterial cells." Annual review of genetics 48 (2014): 537-559.
4.Houseley, Jonathan, and David Tollervey. "The many pathways of RNA degradation." Cell 136.4 (2009): 763-776.
5.Sachs, Alan B. "Messenger RNA degradation in eukaryotes." Cell 74.3 (1993): 413-421.
6.Gallego Romero, Irene, et al. "RNA-seq: impact of RNA degradation on transcript quantification." BMC biology 12 (2014): 1-13.
7.Tan, Siun Chee, and Beow Chin Yiap. "DNA, RNA, and protein extraction: the past and the present." BioMed Research International 2009 (2009).
8.Samadani, Ali Akbar, et al. "RNA Extraction from Animal and Human's Cancerous Tissues: Does Tissue Matter?." International journal of molecular and cellular medicine 4.1 (2015): 54.
9.Jang, Jin Sung, et al. "Comparative evaluation for the globin gene depletion methods for mRNA sequencing using the whole blood-derived total RNAs." BMC genomics 21 (2020): 1-9.
10.Krjutškov, Kaarel, et al. "Globin mRNA reduction for whole-blood transcriptome sequencing." Scientific reports 6.1 (2016): 31584.
11.Kemfack, A.M., Hernandez-Morato, I., Moayedi, Y. et al. An optimized method for high-quality RNA extraction from distinctive intrinsic laryngeal muscles in the rat model. Sci Rep 12, 21665 (2022). https://doi.org/10.1038/s41598-022-25643-y
