在品味葡萄酒的醇香之余,你是否好奇过不同葡萄酒之间究竟隐藏着怎样的秘密?今天,我们将借助Python编程语言和K-means聚类分析模型,通过聚类分析,我们希望能够发现不同葡萄酒之间的相似性和差异性,从而更深入地理解葡萄酒的品质与特性。
K-means模型通过将数据集中的样本划分为若干个簇,使得同一个簇内的样本相似度高,而不同簇之间的样本相似度低,这种算法的核心思想是以空间中点的中心来代表一个簇,并通过最小化每个点到其簇中心的距离之和来寻找最优的簇划分。
1.导入数据首先将读取上传的葡萄酒数据,然后进行初步的探索,以便了解数据的结构和内容。
import pandas as pd# 导入 pandas 作为 pdimport numpy as np # 导入 numpy 作为 np# 导入数据wine_data = pd.read_excel('葡萄酒.xlsx')# 数据预览wine_data.head()
数据集包含以下字段,包含每一个葡萄酒样本的指标数据。
fixed acidity:固定酸度volatile acidity:挥发性酸度citric acid:柠檬酸residual sugar:残糖chlorides:氯化物free sulfur dioxide:游离二氧化硫total sulfur dioxide:总二氧化硫density:密度pH:pH值sulphates:硫酸盐alcohol:酒精name:名称(可能是样本的编号)2.数据预处理接下来,进行数据预处理,包括去除非数值特征和标准化数值特征,为K-means聚类分析做好准备。
from sklearn.preprocessing import StandardScaler # 从 sklearn.preprocessing 导入 StandardScaler,用于数据标准化# 移除非数值列wine_numeric_data = wine_data.select_dtypes(include=[np.number])# 对数据进行标准化scaler = StandardScaler()scaled_data = scaler.fit_transform(wine_numeric_data)3.K-means聚类使用K-means算法对数据进行聚类,并绘制树形图和可视化聚类结果。并且,计算出不同聚簇数量所对应的轮廓系数,以评估聚类的效果。
# 从 sklearn.cluster 导入 KMeans,用于执行 K-means 聚类from sklearn.cluster import KMeans # 从 sklearn.metrics 导入 silhouette_score,用于计算轮廓系数from sklearn.metrics import silhouette_scoreimport matplotlib.pyplot as plt # 导入 matplotlib.pyplot 用于绘图plt.rcParams['font.sans-serif']=['SimHei'] #用来正常显示中文标签plt.rcParams['axes.unicode_minus']=False #用来正常显示负号import numpy as np # 导入 numpy 作为 np# 创建一个列表,用于存储不同聚类数量的轮廓系数silhouette_scores = []# 尝试的聚类数量范围cluster_range = range(2, 11)# 对每个聚类数量执行 K-means 聚类并计算轮廓系数for n_clusters in cluster_range: kmeans = KMeans(n_clusters=n_clusters, random_state=42) cluster_labels = kmeans.fit_predict(scaled_data) silhouette_scores.append(silhouette_score(scaled_data, cluster_labels))# 绘制轮廓系数图plt.figure(figsize=(9, 6))plt.plot(cluster_range, silhouette_scores, marker='o')plt.title('不同聚类数量的轮廓系数')plt.xlabel('聚类数量')plt.ylabel('轮廓系数')plt.grid(True)plt.show()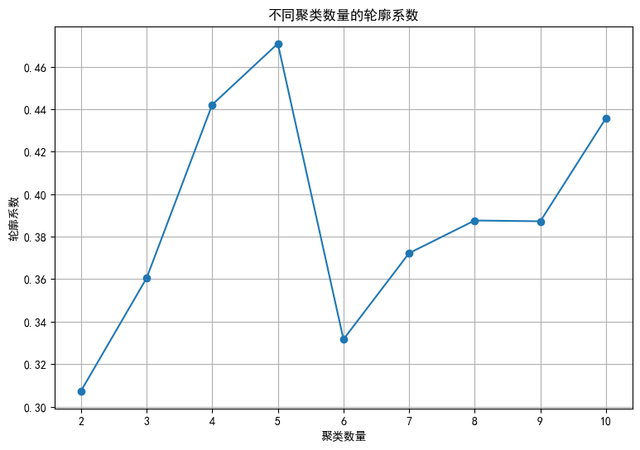
基于轮廓系数找到最佳的聚类数量,并且显示带有聚类标签的数据框的前几行和最佳聚类数量。
# 基于轮廓系数找到最佳的聚类数量best_n_clusters = cluster_range[np.argmax(silhouette_scores)]# 使用最佳的聚类数量执行 K-means 聚类best_kmeans = KMeans(n_clusters=best_n_clusters, random_state=42)best_cluster_labels = best_kmeans.fit_predict(scaled_data)# 将聚类标签添加到原始数据框中wine_data['Cluster'] = best_cluster_labels# 显示带有聚类标签的数据框的前几行和最佳聚类数量print('显示带有聚类标签的数据框的前几行和最佳聚类数量')print('----'*20)print(wine_data.head(), best_n_clusters)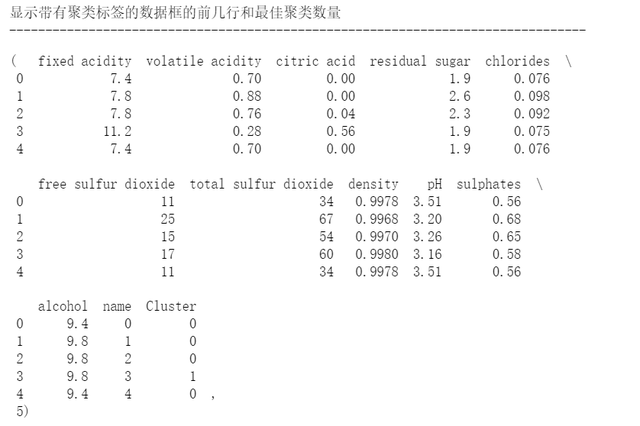
根据轮廓系数图,可以看到当聚簇数量为5时,轮廓系数达到最大值,这意味着最佳的聚簇数量为5,现在,对于这5个聚簇可视化聚类结果,并绘制树形图,直观地展示聚类结果。
#使用二维PCA图可视化聚类结果from sklearn.decomposition import PCAimport seaborn as sns#为可视化降低数据的维度pca = PCA(n_components=2)pca_data = pca.fit_transform(scaled_data)#创建一个用于可视化的数据框pca_df = pd.DataFrame(data=pca_data, columns=['PC1', 'PC2'])pca_df['Cluster'] = best_cluster_labels#按聚类绘制PCA结果plt.figure(figsize=(9, 6))sns.scatterplot(data=pca_df, x='PC1', y='PC2', hue='Cluster', palette='viridis', legend='full')plt.title('葡萄酒数据的PCA图,按聚类着色')plt.xlabel('主成分1')plt.ylabel('主成分2')plt.show()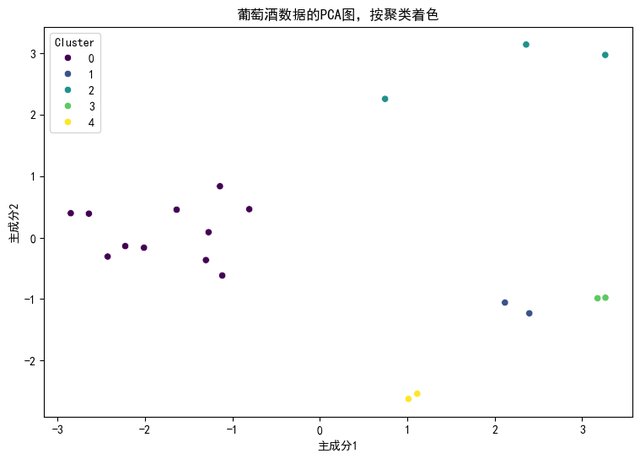
上图展示了使用主成分分析(PCA)将数据降至二维后的聚类结果。不同的颜色代表不同的聚簇,可以观察到各个聚簇之间的分布情况。
4.树形图作出葡萄酒数据的树形图,它展示了数据点之间的层次关系,根据树形图的形状,可以进一步了解不同聚簇之间的相似性和差异性情况。
从树形图中,可以观察到不同聚簇之间的相似性和差异性,以及它们如何根据特征被逐步合并,使用K-means聚类分析成功帮助我们识别出葡萄酒数据集中的自然分组。
#使用树状图(dendrogram)可视化聚类结果from scipy.cluster.hierarchy import linkage, dendrogram#计算树状图的链接矩阵linkage_matrix = linkage(scaled_data, method='ward')#绘制树状图plt.figure(figsize=(9, 6))dendrogram(linkage_matrix, labels=best_cluster_labels, leaf_rotation=90, leaf_font_size=8)plt.title('葡萄酒数据的树状图')plt.xlabel('样本索引')plt.ylabel('距离')plt.show()
根据轮廓系数图,当聚簇数量为5时,轮廓系数达到最大值,这意味着将数据分为5个聚簇是最合适的。同时,在PCA图中,我们可以看到不同聚簇在二维空间中的分布情况,每个聚簇的数据点都相对集中,表明K-means算法能够有效地将相似的数据点划分为一个聚簇。
通过K-means聚类分析,我们成功地将葡萄酒数据集划分为几个不同的簇,每个簇代表了具有相似特性的葡萄酒群体。这一发现不仅有助于我们更深入地理解葡萄酒的品质与特性,也为葡萄酒的分类和推荐提供了有价值的参考。
