lncRNA产品系列上篇介绍了lncRNA的功能及分类,重点阐述了不同分类的lncRNA的功能特征,以及lncRNA在不同位置的功能异质性,NGS技术的普及,对于lncRNA的检测通量也逐渐提高;本篇根据lncRNA的结构特征,将重点介绍lncRNA的富集和高通量测序流程。
LncRNA的特征
LncRNA为超过200nt的长链非编码转录本,部分lncRNA具有polyA结构,其他没有polyA结构的lncRNA,可以是基因间的、反义的或内含子的,它们通常来源于“假基因”, LncRNA的表达水平较低,但在特定细胞类型或生理状态下可能有特异高表达。
由于部分lncRNA没有明确的可以捕获的结构,而且lncRNA的含量相对mRNA少一些,因此需要进行高效的富集。

LncRNA的占比情况
LncRNA的富集
lncRNA中只有部分具有polyA结构(成熟的mRNA均具有polyA结构),因此不能采用oligo-dT的方式富集lncRNA;需要采用其他手段进行lncRNA的富集用于高通量测序,一般采用的是rRNA-remove的流程去除占比最高的rRNA,然后对剩下的mRNA、lncRNA和circRNA进行建库测序。
1.1 rRNA remove
结合试剂盒进行rRNA的去除,去除rRNA的原理是:针对特定物种的rRNA设计相应的探针,探针与rRNA结合,可以进行定向的Rnase H消化,进而达到去除rRNA的目的。
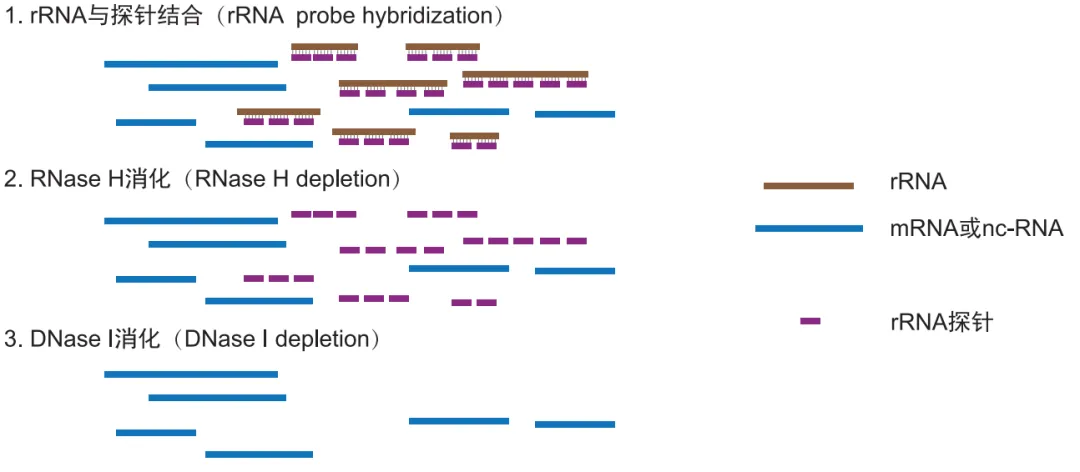
rRNA-remove原理
建库流程
上述经过富集的lncRNA进入文库构建阶段, 需要先经过片段化处理成250bp左右的小片段,反转成cDNA并连接上adaptor,同时为了保证检测到链特异性,需要使用UNG酶消化cDNA二链,进而保证测序可以获得具体的链特异性信息。
LncRNA(也会包括mRNA)构建好的文库结构如下所示:
与flow cell 结合的adaptor(P5P7)+index(标记样品)+测序扩增引物(Rd1Rd2 SP)+插入片段(insert)
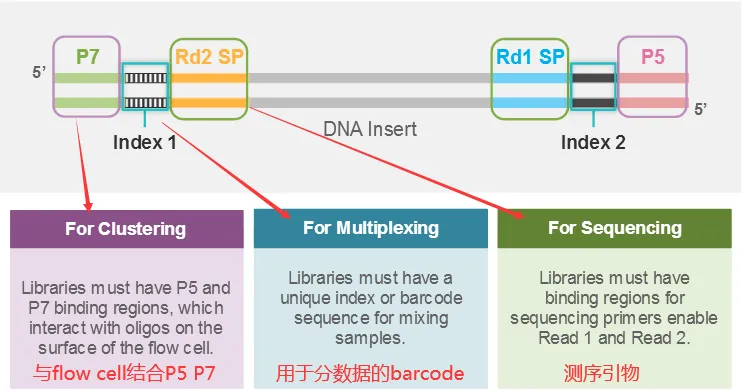
1.1 链特异性建库
RNA-seq建库的时候,直接使用随机引物进行第二条链合成以及加adaptor,这样构建出来的文库就无法识别测序片段式来自于基因组的哪条链上,因为被测序的可能是基因的正义链或者反义链;因此为了区分测序片段(reads)来源(lncRNA既有可能是正义链也有可能是反义链产生的),需要进行链特异性建库,链特异性建库可以更加精准的对lncRNA进行定量。常规采用的链特异性方法是dUTP method:这种方法在合成第二条cDNA链的时候,把dTTP换成dUTP,这样cDNA中原来T的位置全部变成了U,可以进行USER酶特异性降解带有U的那条链,进而保证检测的链特异性。
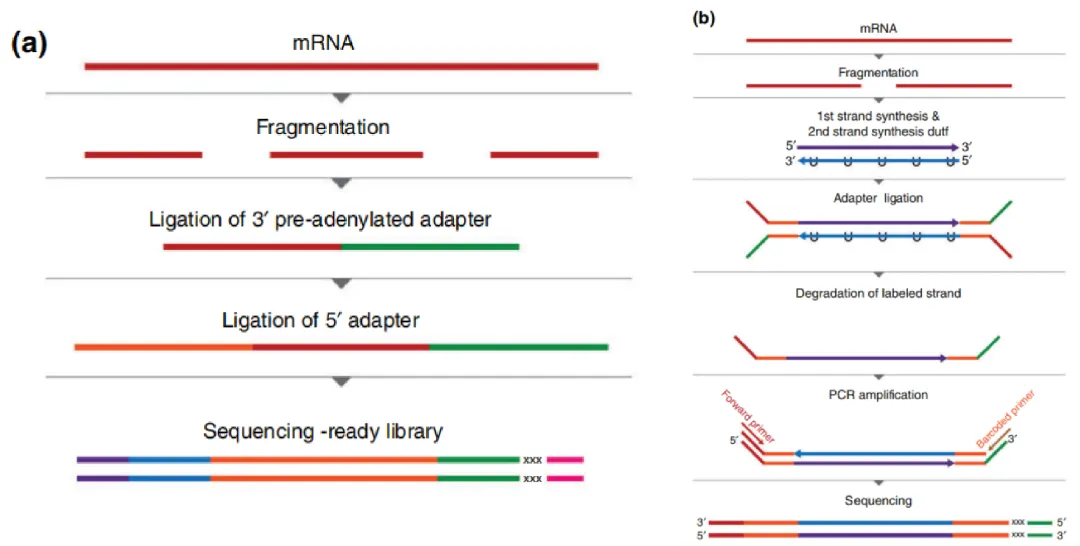
非链特异性建库和链特异性建库
上机测序
由于进行rRNA-remove后得到的RNA中主要是mRNA和lncRNA,其中mRNA占据较多的量,因此测序的时候为了尽可能多的检测到lncRNA,lncRNA-seq的数据量一般都相对较高,例如进行人类细胞的lncRNA测序的数据量为12G,才能达到lncRNA检测的饱和度。
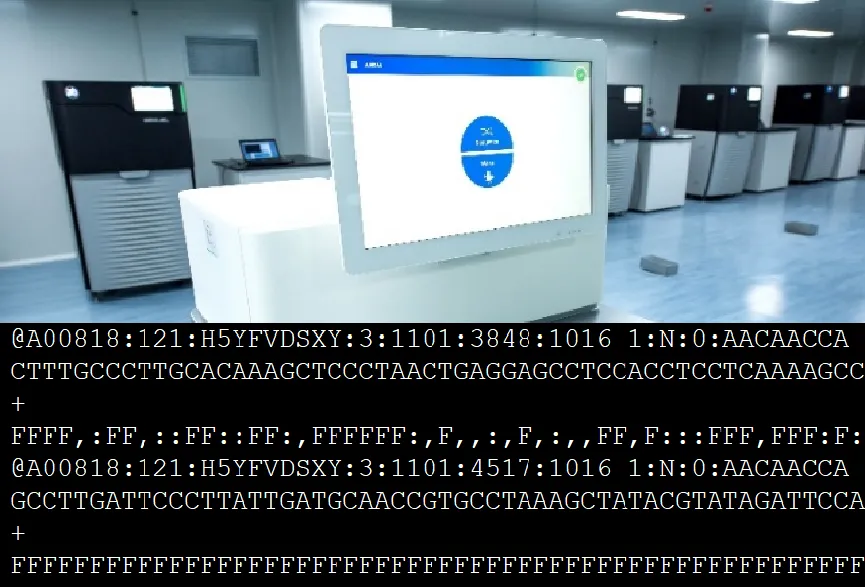
上机平台多采用Novaseq平台,得到的Raw data格式如下,获取数据后可进行后续的数据预处理和相应的lncRNA生信分析。
参考文献
Mattick, John S., et al. "Long non-coding RNAs: definitions, functions, challenges and recommendations." Nature reviews Molecular cell biology 24.6 (2023): 430-447.
Hrdlickova, Radmila et al. “RNA-Seq methods for transcriptome analysis.” Wiley interdisciplinary reviews. RNA vol. 8,1 (2017): 10.1002/wrna.1364. doi:10.1002/wrna.1364
