导语
自上个世纪七十年代第一代Sanger测序技术问世以来,经历了多代测序技术的变革。尽管一代测序作为测序黄金标准,但其数据产出低通量性制约了基因组学的快速发展。因此,新一代测序技术(Next generation sequencing,NGS)应运而生,革命性的提高了测序通量和时效性,推动了多组学研究的大众化。目前主流的二代测序技术是Illumina(因美纳)技术(https://www.illumina.com.cn/)和DNBSEQ(华大智造)技术(https://www.mgi-tech.com/)。那么这两种技术之间究竟什么样的区别呢?我们今天将从多个方面进行深度剖析他们之间的差异~~
测序原理ORIGINGENE
结合Illumina和BGI官方发布的测序原理来看,二者在文库制备、文库扩增以及测序阶段都有差别。让我们来逐一学习并分析~~
(1)文库构建
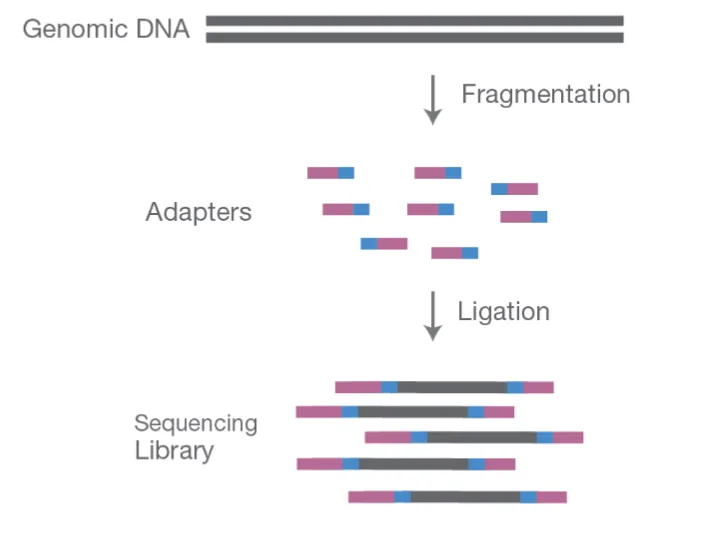
1)Illumina文库构建步骤
A. DNA片段化:
使用机械(如声波破碎)或酶法(如限制性内切酶)将基因组DNA片段化。
B. 末端修复和加A尾:
片段化的DNA末端需要进行末端修复以生成平末端,然后添加A尾形成粘性末端以便于接下来接头的连接。
C. 接头连接:
将含有测序引物和条形码序列(用于样本多重化)的接头连接到片段化的DNA上。
D. PCR扩增:
对连接接头的DNA片段进行PCR扩增,以获得足够量的DNA文库,同时引入测序所需的序列。
E. 文库质控和定量:
使用Qubit、Bioanalyzer等工具对文库进行质控和定量,以确保文库质量和浓度符合测序要求。
Illumina官网
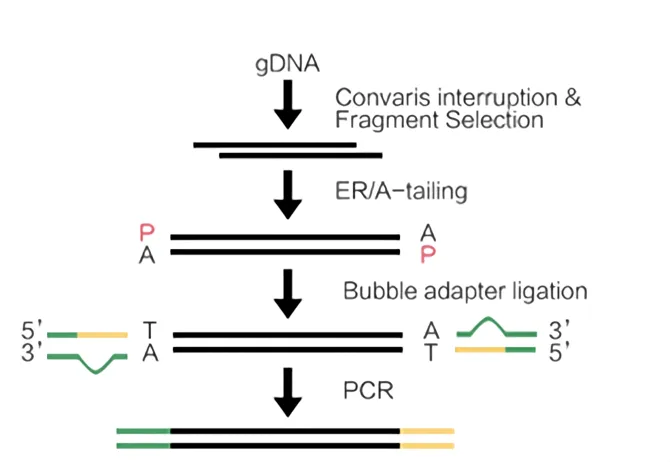
2)BGI文库构建步骤
A. DNA片段化:
与Illumina相似,BGI也使用机械或酶法将基因组DNA片段化。
B. 末端修复和接头连接:
片段化的DNA进行末端修复,然后直接连接特定的Bubble接头,这些接头包含测序引物和条形码序列。
C. PCR扩增
对连接接头的DNA片段进行PCR扩增,以获得足够量的DNA文库。
D. 文库质控和定量:
使用类似的方法对文库进行质控和定量,以确保文库质量和浓度符合测序要求。
华大智造官网
从上述的文库构建来看,不难看出,BGI和Illumina平台在建库阶段最大的区别在末端修复和接头连接时的不同。主要区别在以下三个方面:
1)Illumina文库接头是完全双链的DNA分子,具有已知序列,可以与目标DNA片段通过连接酶反应进行连接;而BGI的Bubble接头由部分双链和部分单链DNA结构组成,在适当的温度条件下形成稳定的双链结构,可以与目标DNA片段高效连接。
2)Illumina的接头设计为平末端连接,这意味着目标DNA片段需要通过末端修复步骤将所有DNA片段修整为平末端,以便与接头连接;而BGI的接头一端具有单链悬垂末端,使其能够与DNA片段的互补序列进行高效连接,减少非特异性连接的发生。
3)Illumina的接头设计主要用于Illumina的测序平台,与其他平台的兼容性较低;而BGI的接头则可以兼容Illumina和BGI。
(2)文库扩增
实际上,Illumina及BGI技术最大的不同在于文库扩增的原理,众所周知,Illumina平台采用的是桥式PCR扩增技术,而BGI则是滚环扩增技术,让我们来具体看看这两种技术的文库扩增流程~~
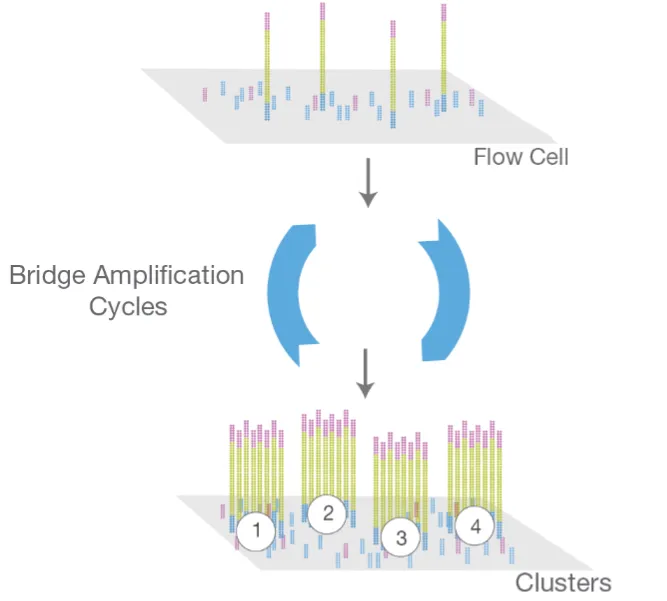
1)Illumina桥式PCR扩增步骤
A.FlowCell(流动池):FlowCell是有着多个lane(泳道)的玻璃板,每个lane可以测一个样本或多样本的混合物,且随机布满了能够与文库两端接头分别互补配对或一致的寡核苷酸(oligos,P7和P5接头)。
B.第一链合成:将双链DNA文库变性解链成单链,当单链DNA模板流过每条泳道时,会被对应的接头捕获,被固定在芯片上,加入dNTP和聚合酶后,以原始模板链为模板,FlowCell上的接头为引物,合成第一链。
C.桥式PCR:加入碱性NaOH溶液,使DNA变性成单链,洗去原始模板链,新合成的链留在FlowCell上,加入中性液体中和,单链DNA弯曲,其顶部接头的序列与附近的oligos互补配对形成“单链桥”,加入DNA聚合酶、dNTP,合成互补链,形成“双链桥式结构”。变性双链的桥,得到与FlowCell相连的两条互补的单链DNA分子,单链DNA分子再一次弯曲,与附近的oligos互补配对形成桥式结构,以此不断重复便可完成DNA的扩增。这个过程称为“桥式PCR扩增”。桥式PCR扩增不断进行,直至形成足够的桥式DNA,形成Clusters。然后通过切断引物的特定基团,加入NaOH溶液解开双链桥式结构并选择性切除反向链,留下正向链,并封闭所有DNA分子的3'端,准备测序。
Illumina官网
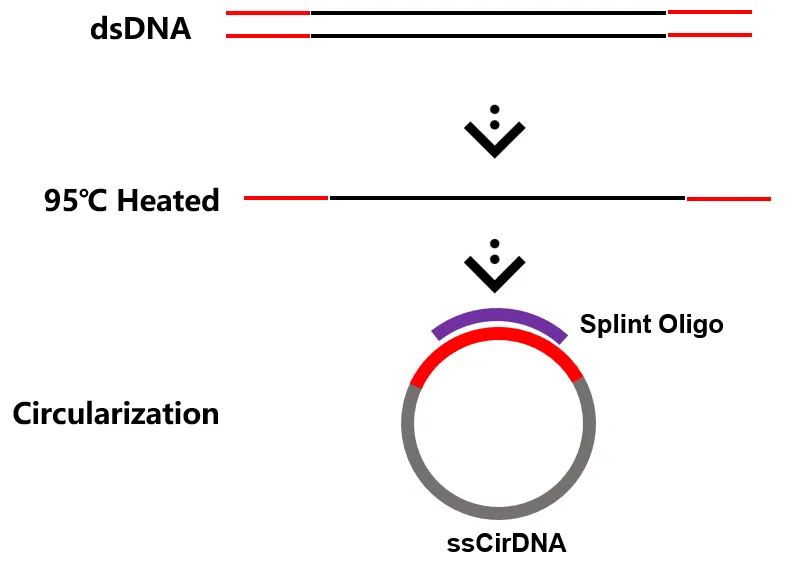
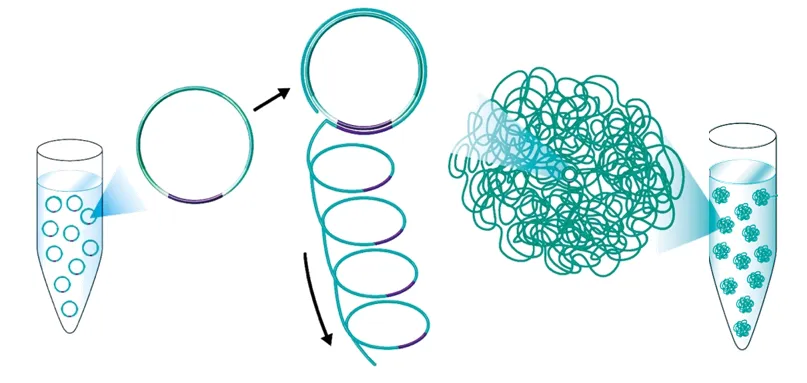
2) BGI滚环扩增步骤
与Illumina不同,BGI的文库扩增步骤包括环化DNA片段,然后通过滚环扩增(Rolling Circle Amplification,RCA)生成大量的DNA纳米球(DNB)。这些DNB含有大量的DNA拷贝,便于在测序过程中进行高效的信号检测。
A.DNA单链环化:将带有接头序列的双链DNA(Double-stranded DNA,dsDNA),通过高温变性形成单链DNA(Single-stranded DNA,ssDNA),环化引物与ssDNA的两端互补配对,在连接酶的催化下,ssDNA的首尾相连接,形成单链环状DNA(Simgle-strand circular DNA,sscirDNA)。
华大智造官网
B.DNB制备:以单链环状DNA为模板,在DNA聚合酶作用下进行滚环扩增(RCA),将单链环状DNA扩增到100-1000拷贝,扩增产物称为DNB。DNB通过简单的质量浓度质控后,就可以用于下一步的上机测序,操作简单,且不需要昂贵的定量设备和耗材。
基于RCA的线性扩增技术,每次扩增都是以原始的DNA单链环为模板,使用保真性极高的聚合酶,使得在DNB的所有拷贝的同一个位置上出同样的错的几率几乎是零。RCA扩增技术有效的避免了PCR扩增错误指数积累的问题,从而大大提高测序的准确性。
华大智造官网
在文库扩增阶段,Illumina和BGI平台的主要差别就在于Illumina使用传统的PCR扩增方法生成线性DNA拷贝,而BGI则采用滚环扩增(RCA)技术生成DNA纳米球(DNB)。这一扩增技术的差异不仅影响文库的扩增效率和误差率,也对后续的测序性能和数据质量产生重要影响。BGI的DNA纳米球技术通过生成高拷贝的DNA片段,提高了测序信号的强度和稳定性,成为了其在测序市场上的一大优势。
(3)上机测序
最后就是测序上机测序阶段啦~~这里Illumina平台采用的是边合成边测序技术(SBS)原理,华大则主要是联合探针锚定聚合技术(cPAS)。
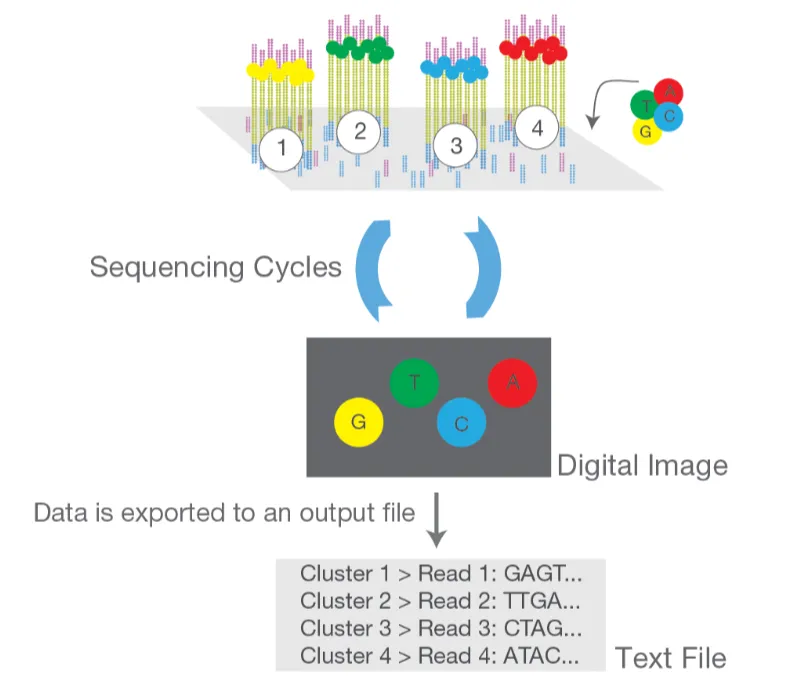
1)Illumina上机测序步骤
A. 引物延伸:加入测序引物,使其结合至测序引物结合位点。
B. 荧光标记核苷酸的加入:加入改造过的DNA聚合酶和带有4种荧光标记的dNTP,这些核苷酸是“可逆终止子”,其3’末端连接的是叠氮基团,使得每个循环只能延长单个碱基。
C. 信号检测:高分辨率相机检测每个克隆群发出的荧光信号,记录下每个碱基的位置。
D. 去除终止剂和荧光标记:去除核苷酸上的终止剂和荧光标记,使下一个核苷酸可以继续加入。
E. 循环重复:重复引物延伸、荧光标记核苷酸加入、信号检测和去除标记的过程,直至读取完所有的DNA序列。
F. 双端测序:在读取正向DNA链后,洗脱产物,继续读取反向DNA链的过程。
Illumina官网
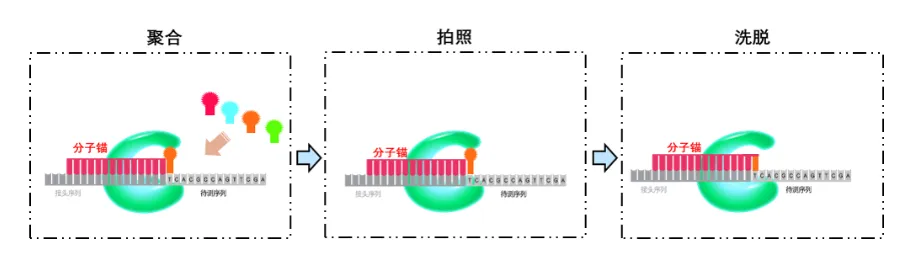
2) BGI上机测序步骤
A. DNB加载:DNB在酸性条件下带负电,在表面活化剂的辅助下,通过正负电荷的相互作用,被加载到规则阵列载片中有正电荷修饰的活化位点,每个纳米孔中可以容纳一个或多个DNA纳米球。
B. cPAS技术:在DNA聚合酶的催化下,将测序引物锚定分子和荧光探针在DNB上进行聚合,洗脱掉未结合的探针后,在激光的作用下荧光信号被激发,随后利用高分辨率成像系统对光信号进行采集、读取和识别,从而获得当前待测碱基的序列信息,然后加入再生洗脱试剂,去除荧光基团,进入下一个循环的检测。
华大智造官网
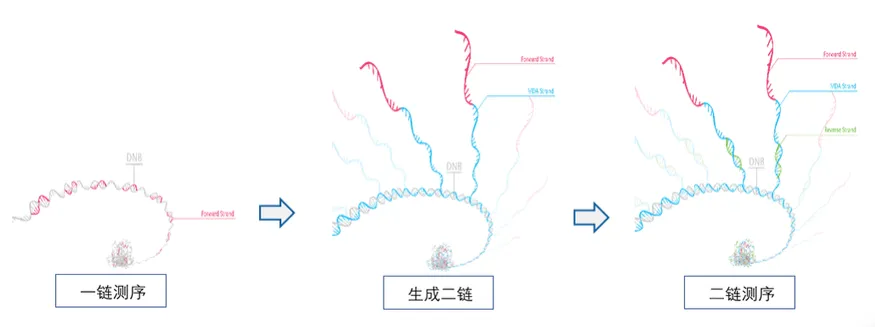
C. 二链测序:在完成一链测序后,加入具有链置换功能的DNA聚合酶进行DNA二链合成反应,在持续的延伸过程中,当遇到双链结构的时候,在DNA聚合酶的解旋作用下,完成边解旋边复制的反应,形成大量的单链DNA作为二链测序的模板,然后杂交二链测序引物,开始二链的cPAS测序。利用DNA聚合酶的链置换特性,实现了DNB的双端测序,而且重新合成的二链拷贝数更多,能够获得更强的荧光信号,有效的提高了测序的准确性。
华大智造官网
主流产品型号及产出量ORIGINGENE
当前主流的二代平台包括Illmina的NovaSeq 6000、NextSeq 2000和NovaSeq X plus以及BGI的DNBSEQ-T7和DNBSEQ-G99,这些不同型号的测序平台在最小读长、最大读长、最大通量以及运行时间等方面有一些差异,但是测序数据质量及可用性相当。
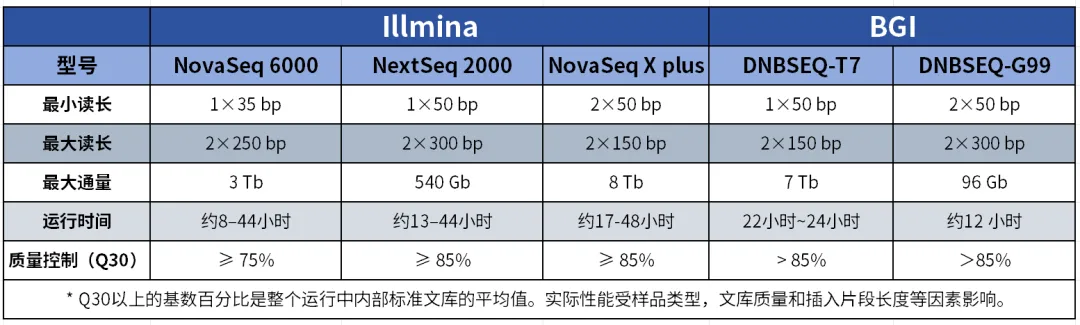
注:资料来自于Illumina及BGI官网。
以上我们主要了解了Illumina和BGI平台在文库构建、扩增以及测序技术上的主要原理和主流产品产出量的差异,那么在实际应用中,这两种测序平台表现又是如何呢?且看下回分解~
