导语
上期,我们一起深入剖析了Illumina和BGI的不同平台在通量、读长以及测序原理等方面的差异(详情请点击链接:技术介绍 | BGI与Illumina的前世今生(一))。虽然这两种技术在原理方面的差异甚大,但是它们在市场上的使用率都是十分之高,那么他们在实际不同的测序应用中是否有明显的测序质量及项目兼容性方面的差异呢?让我们从几篇文献中一起看看这两种平台的在不同测序项目中的表现吧~
全基因测序
文献标题:Comparison between MGI and Illumina sequencing platforms for whole genome sequencing

主要研究方法及结果:该研究使用Illumina的NovaSeq 6000和MGI的MGISEQ-2000以及DNBSEQ-T7三种平台对两对来自韩国肺癌患者的正常组织和肿瘤组织进行了测序。然后,分别对体细胞和生殖系的单核苷酸变异(SNVs)和插入缺失(indels)进行了检测,以比较这三个平台之间的性能。在质控分析中,三个平台均显示出高质量得分和深覆盖度。三者比较显示,MGISEQ-2000在胚系SNVs和indels检测方面与NovaSeq 6000最为一致,而DNBSEQ-T7在体细胞SNVs和indels检测方面与NovaSeq 6000最为一致。这些结果表明,MGISEQ-2000和DNBSEQ-T7平台的性能可与Illumina的NovaSeq 6000平台媲美,并支持MGISEQ-2000和DNBSEQ-T7平台在实际基因组分析领域的潜在适用性。
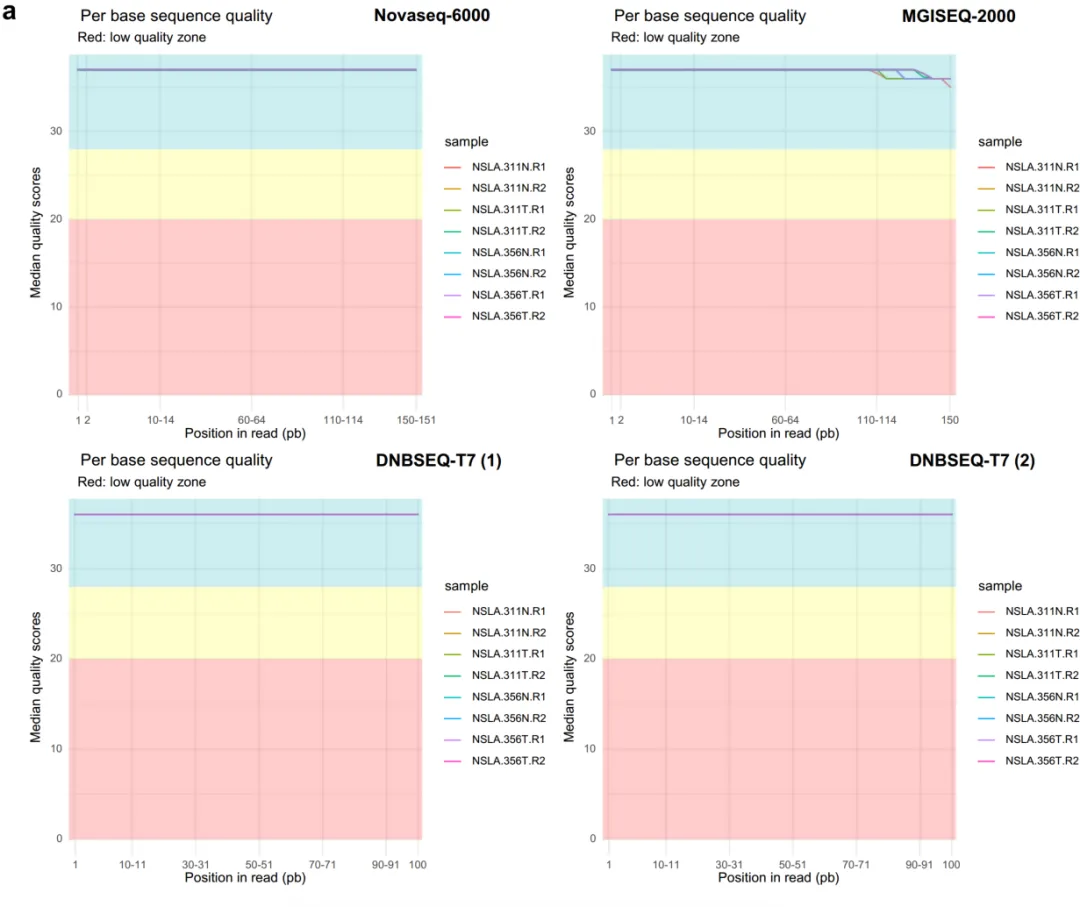
图1 三个测序平台的FastQC结果。a表示NovaSeq 6000、MGISEQ-2000、DNBSEQ-T7(1)、DNBSEQ-T7(2)的总单碱基序列质量,b表示NovaSeq 6000、MGISEQ-2000、DNBSEQ-T7(2)的总单序列GC含量。
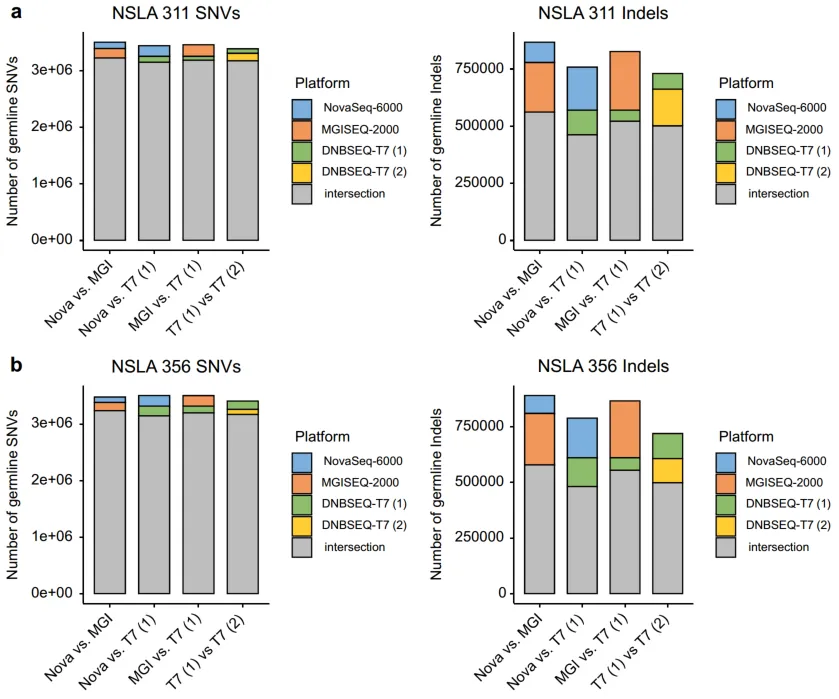
图2 胚系SNVs和indels的比较。展示了变异重叠的数量。重叠的变异(交集)以灰色显示。
微生物宏基因组测序
文献标题:Assessment of the cPAS-based BGISEQ-500 platform for metagenomic sequencing

主要研究方法及结果:该研究使用20名健康个体的粪便样本,在BGISEQ-500平台上进行了包含8个文库重复和8个测序重复的设置,评估了其在宏基因组测序中的平台内重现性。通过比较BGISEQ-500平台与Illumina HiSeq 2000平台和Illumina HiSeq 4000平台的20对平行重复,评估了跨平台的一致性。此外,还比较了两个Illumina平台彼此之间的性能。通过新开发的整体准确性质量控制方法,BGISEQ-500平台每个样本平均获得82.45百万条高质量读数(占原始读数的96.06%),其中90.56%的碱基得分为Q30及以上。定量分析显示,BGISEQ-500平台内重复之间具有极高的再现性。跨平台重复的差异略大于平台内重复,但仍表现出高度一致性。仅有低百分比(2.02%–3.25%)的基因在BGISEQ-500与HiSeq平台相比时相对丰度存在显著差异,且HiSeq平台更容易富集高GC含量的基因。该研究首次提供了使用BGISEQ-500进行人类肠道宏基因组测序数据的性能指标。高准确性和技术重现性确认了新平台在宏基因组研究中的适用性。
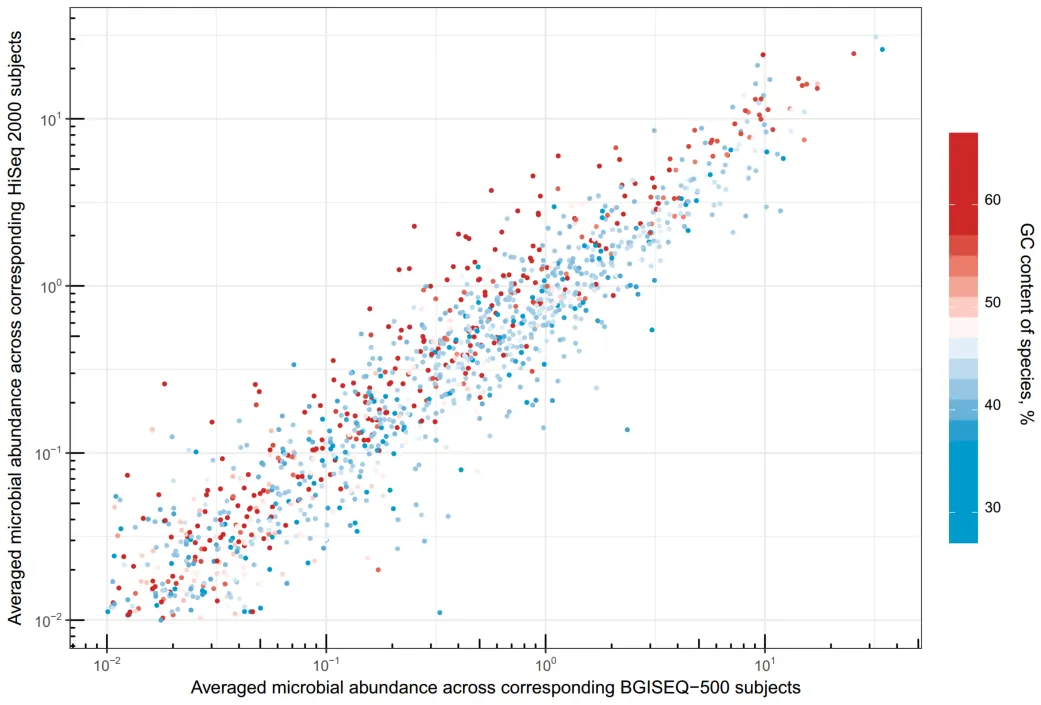
图1 BGISEQ-500与HiSeq 2000相对物种丰度的比较
转录组测序
文献标题:Comparative performance of the BGISEQ-500 and Illumina HiSeq4000 sequencing platforms for transcriptome analysis in plants

主要研究方法及结果:该研究针对BGISEQ-500和Illumina HiSeq4000之间的跨平台比较研究,分析了拟南芥(Arabidopsis thaliana)野生型(Col 0)的转录组。在生物重复实验中,使用上述平台评估RNA测序和转录组数据处理的关键参数。BGISEQ-500和Illumina HiSeq4000这两个平台的结果在基因定量、差异表达基因的鉴定和可变剪接事件的检测方面显示了高度一致性,跨平台(相关性0.88–0.93)和平台内(相关性0.95–0.98)比较都表现出高相关性。该案例提供了一个全面的参考数据集,以验证BGISEQ-500的能力,使其确立为一个有竞争力和可靠的植物转录组测序平台。
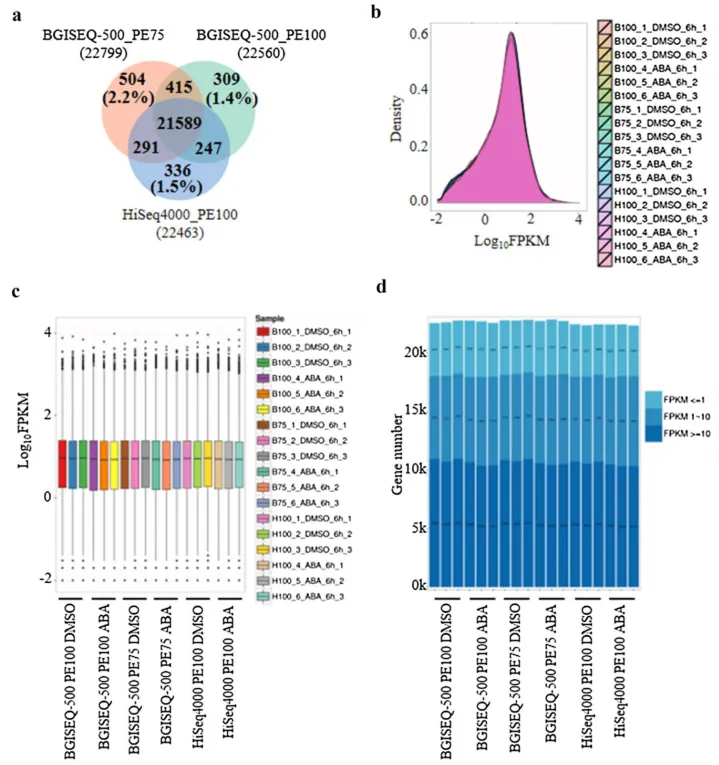
图1 三种测序方法中基因检测和定量的重复性
单细胞转录组测序
文献标题:Comparative performance of the BGI and Illumina sequencing technology for single-cell RNA-sequencing

主要研究方法及结果:高通量单细胞RNA测序(scRNA-seq)平台(如10x Genomics的Chromium)生成的文库通常需要大量的测序,这主要是由于细胞数量庞大。数据质量直接影响到使用这些数据解决生物学问题的能力。该研究使用由10x Genomics Chromium平台生成的包含超过70,000个细胞的相同单细胞文库,对Illumina NextSeq 500、NovaSeq 6000与MGISEQ-2000平台的性能进行了比较。结果表明,NovaSeq 6000和MGISEQ-2000在测序质量、基因检测、细胞条形码和独特分子标识符检测方面具有高度可比的性能。NextSeq 500在相同指标上的性能也与MGISEQ-2000相似。两种测序平台生成的数据在一般单细胞分析中的结果非常相似。此外,NextSeq 500和MGISEQ-2000在通过变异检测进行多重细胞池解卷积以及检测来自汇集CRISPR单细胞筛选的引导RNA(gRNA)方面的性能也具有可比性。该研究为应用于高通量单细胞RNA测序文库的高通量测序平台提供了基准。
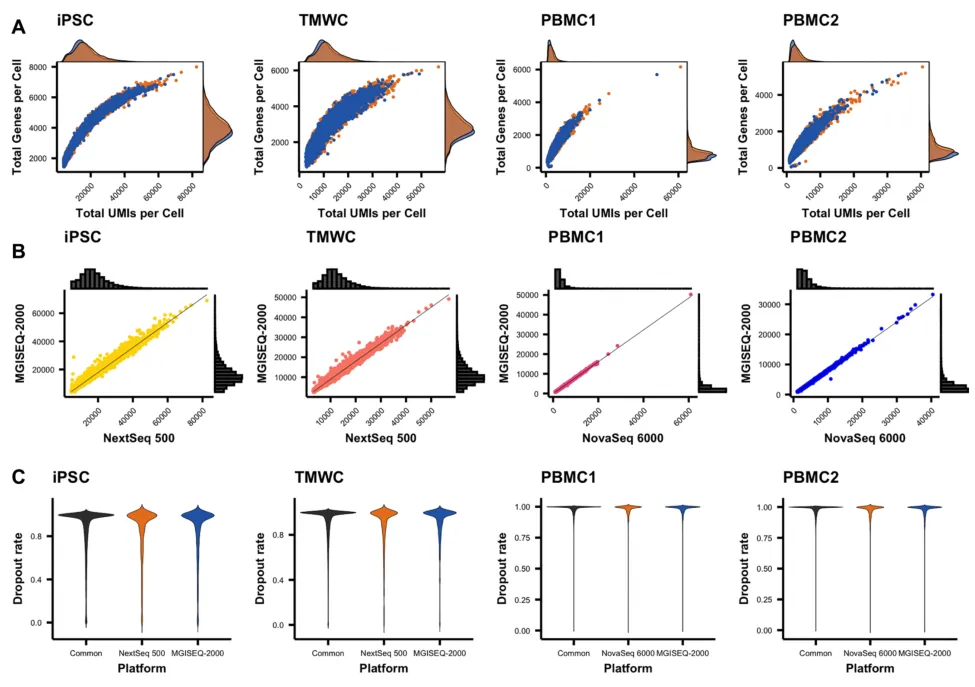
图1 平台检测到的细胞和基因。两种技术在检测细胞和基因方面表现出相似的灵敏度.(A)各平台捕获效率;(B)细胞中检测到的分子总数;(C)平台检测的基因间丢失率。
参考文献
[1] Jeon, S. A., Park, J. L., Park, S. J., Kim, J. H., Goh, S. H., Han, J. Y., & Kim, S. Y. (2021). Comparison between MGI and Illumina sequencing platforms for whole genome sequencing. Genes & Genomics, 43, 713-724.[2] Fang, C., Zhong, H., Lin, Y., Chen, B., Han, M., Ren, H., ... & Li, J. (2018). Assessment of the cPAS-based BGISEQ-500 platform for metagenomic sequencing. Gigascience, 7(3), gix133.[3] Zhu, F. Y., Chen, M. X., Ye, N. H., Qiao, W. M., Gao, B., Law, W. K., ... & Liu, Y. G. (2018). Comparative performance of the BGISEQ-500 and Illumina HiSeq4000 sequencing platforms for transcriptome analysis in plants. Plant methods, 14, 1-14.[4] Senabouth, A., Andersen, S., Shi, Q., Shi, L., Jiang, F., Zhang, W., ... & Powell, J. E. (2020). Comparative performance of the BGI and Illumina sequencing technology for single-cell RNA-sequencing. NAR genomics and bioinformatics, 2(2), lqaa034.